- Accueil
- D.Sciences biologiques
- II.Biologie moléculaire COMPLET
- Bio moléculaire 9
Bio moléculaire 9
II.7 Traduction ou synthèse des protéines (protéogénèse)
III.7.1 Le code génétique
L’ARN est une copie aussi fidèle que possible de l’ADN, il porte son information au niveau de quatre bases A U G C.
Comment coder 20 acides aminés différents au moyen de quatre bases différentes ?
Raisonnement :
1 base pour 1 aa : 4 possibilités
Suite de 2 bases pour 1 aa : 42
Suite de 3 bases pour 1 aa : 43
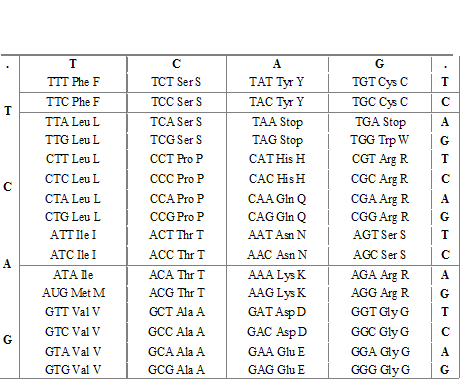
Le code sera formé de 64 triplets ou codons
3.4.2 ) Représentation du code 1 ère forme (ADN codant)
Autre présentation du code génétique , 2ème forme ( ARNm)
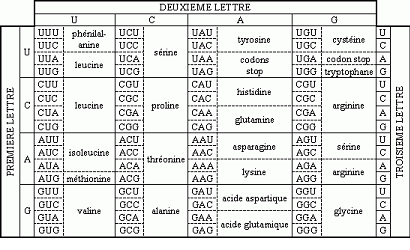
Code des acides aminés ( réciproque du code génétique)
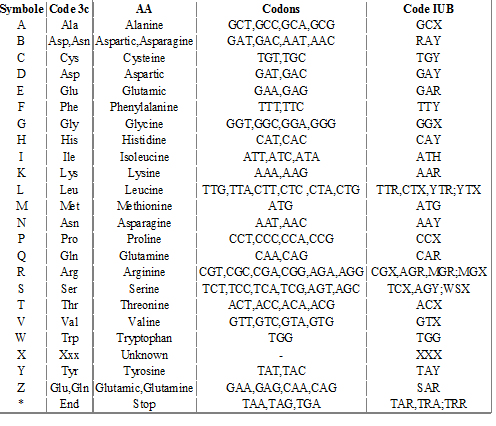
Rappel : à un codon correspond un seul acide aminé, à un acide aminé correspondent plusieurs codons, le code est en quelque sorte non bijectif .
Caractéristiques du code : nous nous baserons sur la seconde forme du code
1° Existence de trois codons non sens
C’est à dire non traduisible en acides aminés : UAA, UAG, UGA.
Ce sont des codons de ponctuation ( fin de traduction).
En ce qui concerne les 61 codons restant : deux aa sont codés par un seul codon, il s’agit de Met et Trp et 18 acides aminés sont codés
par plusieurs codons.
2° Le code est universel : quelques rares exceptions
Mitochondries :
AUA pas Ile mais Met.
UGA pas stop mais Trp. (Mycoplasma capricolum également)
AGA pas Arg mais stop.
AGG pas Arg mais stop.
Paramécies :
UAA et UAG pas stop mais Glt ( Paramécium ).
Levures
3° Le code est dégénéré, redondant
Presque tous les aa sont codés par plusieurs codons
Les codons représentant le même aa ne différent que par la troisième base(lettre), voir les quatre codons de la Sérine.
Cette redondance constitue un système de protection vis à vis des mutations qui peuvent se produire : en effet, les mutations au niveau de la troisième base n’ont pas d’effet.
4° Le code est non chevauchant
Il était dit que le code génétique était non chevauchant, c’à d que la lecture se faisait de la façon
suivante :
UUU/ACG/AUG/UA…………………
1 2 3 4 codons ( cadre de lecture )
Phe Thr Met ………………….
Ce cadre de lecture reste le cadre de base, mais des exceptions sont apparues, notamment chez les Virus.
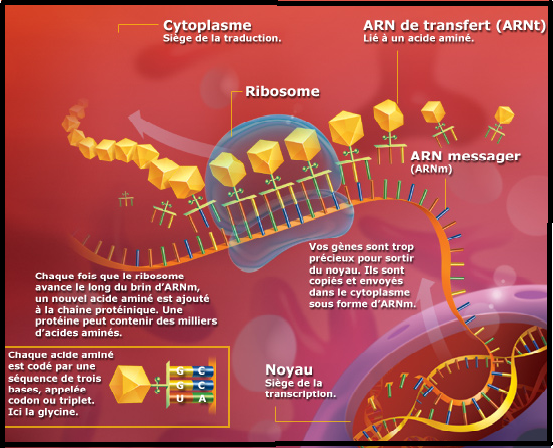
III.7.2 Le lieu de la traduction
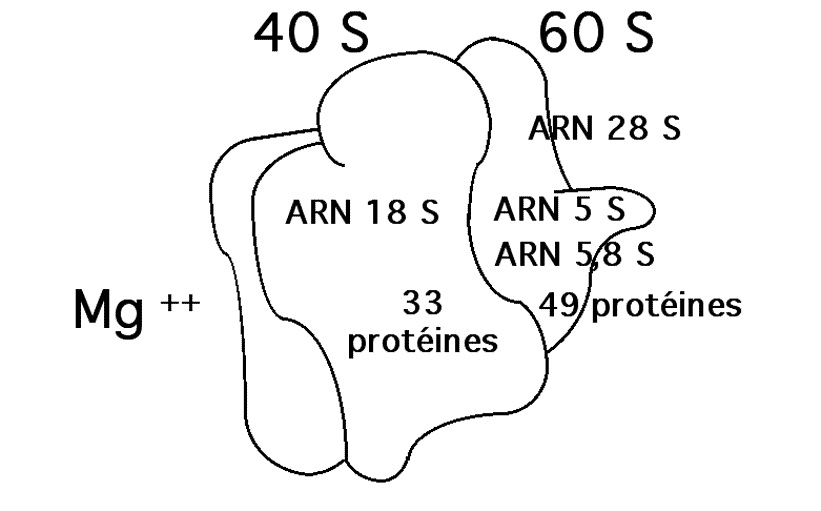
Le lieu de la traduction est dans le cytoplasme, au niveau des ribosomes . Ces ribosomes sont des sites d’accueil pour les ARNm et les ARNt + aa.
Les ribosomes sont constitués comme nous l’avons vu plus tôt d’ acides ribonucléiques et de nombreuses protéines.
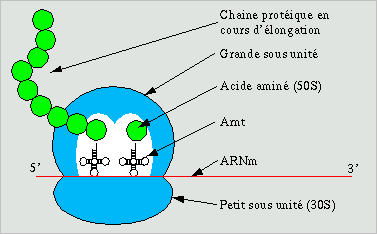
En fait ces ribosomes contiennent deux sites d’accueil pour aa-ARNt (sites P et A)
Parmi ces nombreuses protéines, il y en a une qui joue le rôle d’ « agrafeuse » : elle « agrafe » les aa à la chaîne peptidique. C’est la Peptidyl transférase .
 III.7.3 Les éléments nécessaires à la traduction.
III.7.3 Les éléments nécessaires à la traduction.
- des acides aminés (aa) qui formeront par liaisons peptidiques la structure primaire des protéines.
- ARNm,transcrit final qui apporte l’information.
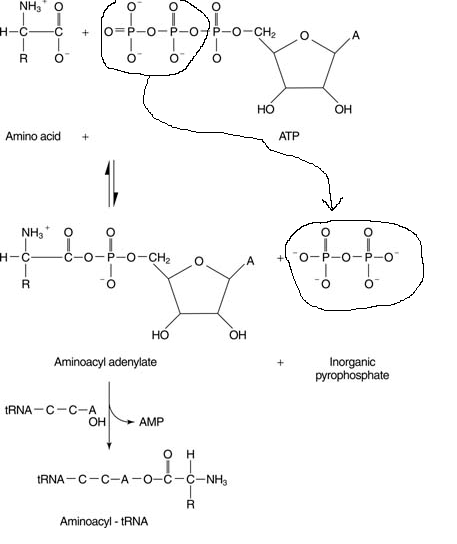
- Les ARNt ou adaptateur entre ARNm et aa, les aa sont fixés sur eux par une liaison ester riche en énergie
aa + ARNt + ATP--------> aa-ARNt + AMP + 2 Pi 
.
III.7.4. Les différentes étapes de la traduction
III.7.4.1 vue globale
III.7.4.2 détails de la traduction.
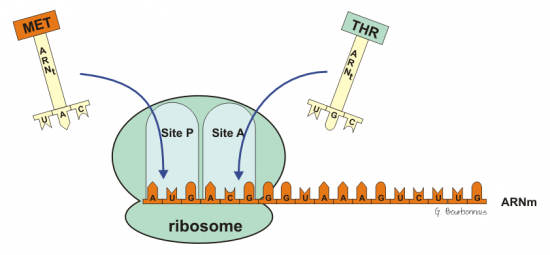
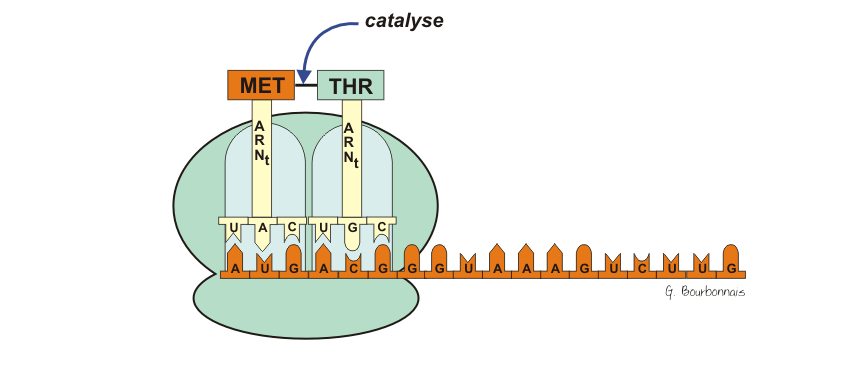
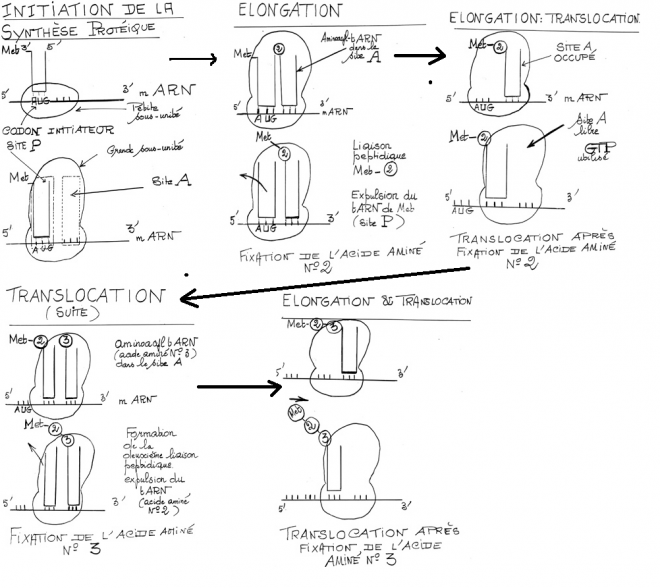
A) L’initiation
la lecture de l’ARNm se fait dans le sens 5’ 3’
Le codon n°1 est le codon initiateur, AUG ------------ Méthionine.
Il existe deux ARNt différents mais ayant le même anticodon pour transporter Met selon qu’il s’agit :
- D’une Met initiale.
- D’une Met destinée à être incorporée en cour de chaîne.
Chez E.coli, la Met initiale n’a pas son groupe NH2 libre, il est bloqué par l’acide formique : formyl méthionine ( f.Met).
ARNt-Met a une conformation qui lui permet d’être logé dans le site peptidique du ribosome.
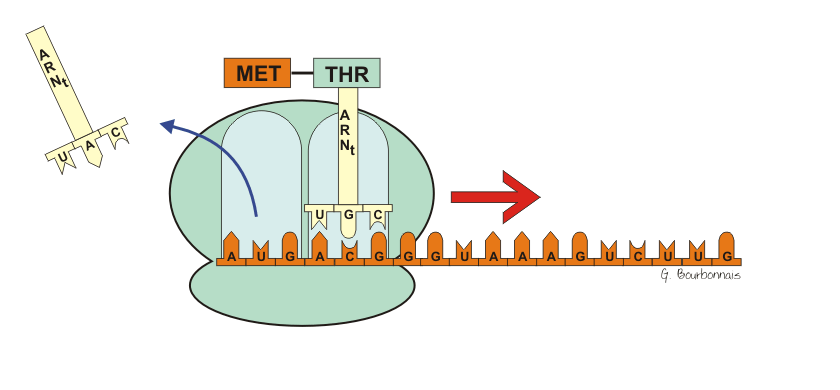
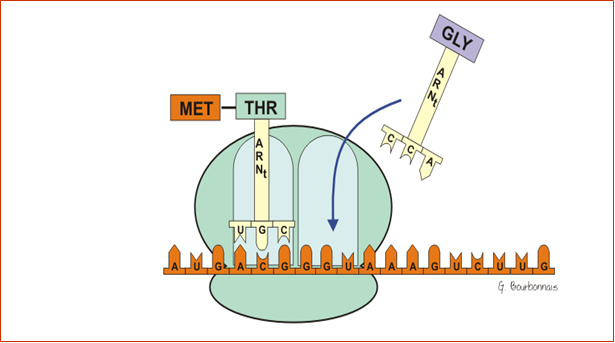
- Accrochage d’un nouveau aminoacyl-ARNt
- Formation de la liaison peptidique
- Translocation du ribosome d’une distance équivalente à un codon.
La protéine est synthétisée depuis l’extrémité N terminale à l’extrémité C terminale.
Le secondARNt peut partir, ensuite réalisation de la liaison peptidique, le ribosome avance et on recommence.
C) Terminaison
- Le dernier codon est toujours un codon ponctuation ou stop. Pas d’ARNt correspondant , ce qui provoque le décrochage de la chaîne polypeptidique.
Une protéine d’un poids moléculaire de 70.000 est formée d’environ 700 aa ( poids moléculaire moyen d’un aa : +/- 100) 700 aa à
assembler …
D) Quelques précisions
a) Revenons à la formation de l’aminoacyl-ARNt
Enzyme : l’aminoacyl-ARNt synthétase est spécifique de l’aa et de l’ARNt correspondant, la sélection de l’acide aminé se fait sur base de :
1) La charge de l’aa
2) L’hydrophobicité de l’aa, avec pour exemple la tyr ARNt synthétase qui distingue immédiatement Tyrosine et Phénylalanine
3) Certains traits structuraux propres ( caractère stérique), rôle important de la tige acceptrice dans l’angle intérieur du L constitué par l’ARNt.
b) Remarques sur les ribosomes
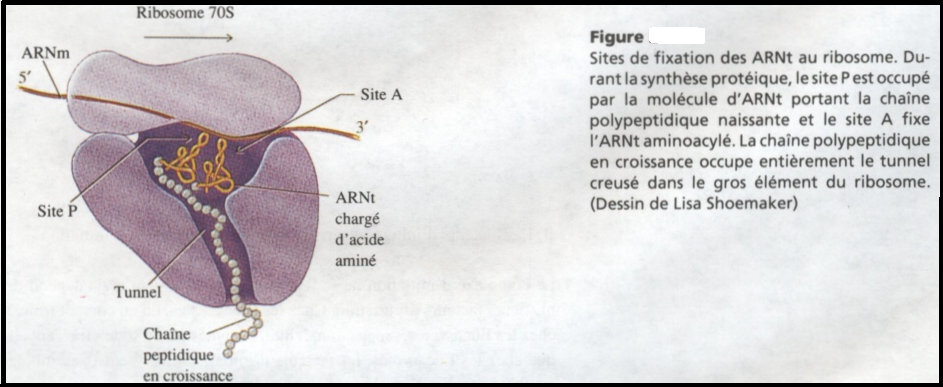
1) Le ribosome fait partie d’un complexe de traduction constitué de l’ARNm,de l’aaARNt, du ribosome et de facteurs protéiques auxiliaires ( facteurs d’’initiation).
2) Le ribosome ménage dans sa structure des sites de formes particulières :
- Un sillon où loge l’ARNm
- Un tunnel de 10 nm de longueur s’ouvrant à l’endroit où se construit la liaison peptidique et qui reçoit la chaîne protéique.
3) Le ribosome possède deux sites de fixation de l’aaARNt.
c) Formation du complexe d’initiation
Le complexe d’initiation assure le bon choix du cadre de lecture et du bon codon d’initiation.
- Présence d’un codon stop ( procaryotes )
- Présence en plus, environ 10 nucléotides avant le codon AUG d’une séquence consensus le précédent 5’ AGGAGG3’ ( séquence de SHINE DALGARNO ) reconnue par la petite sous unité du ribosome ( procaryotes).
- D’autre part, en 5’ du codon initiateur en règle AUG (codant pour la méthionine), il existe une séquence retrouvée dans de nombreux mARN de vertébrés appelée séquence consensus de KOZAK: CCA/GCCAUGG. Cette
séquence facilite l’association entre la petite sous-unité du ribosome et le mARN. Cette séquence consensus comporte le premier AUG, qui est le codon le codon initiateur le plus fréquent chez les eucaryotes.
La formation du complexe d’initiation dépend de l’intervention de plusieurs facteurs d’initiation :
- Au nombre de trois chez les procaryotes ( IF-1, IF-2, IF-3 ).
- Au nombre de huit chez les eucaryotes ( sigle e-IF )
Ils catalysent l’assemblage de l’appareil de synthèse protéique au niveau du codon d’initiateur.
d) Formation de la liaison peptidique catalysée par la peptidyl transférase , et grâce à l’énergie apportée par la liaison ester de l’aaARNT.
e) Facteurs de largage
Exemple chez E.coli : RF-1, RF-2,RF-3.
Les codons fin au site A du ribosome ne sont pas reconnus par un ARNt
(UGA, UAG, UGA ). Le site A va être occupé par RF-3, qui lui même favorisera la fixation au même site de RF 1 ou RF2 , en fonction du codon stop présent. Après fixation de RF1 ou de RF 2, il devient possible pour RF-3 d’hydrolyser un GTP en GDP , ce qui stabilise la fixation de RF 1 ou RF 2.
F ) Bilan énergétique
1) assemblage du complexe d’initiation : GTP ---------> GDP +Pi + E1.
2) Formation de la liaison aa-ARNt riche en énergie : ATP ---------> AMP + 2Pi +E2
3) Positionnement de ARNt-aa au site A : GTP ---------->GDP + Pi +E3
4) Translocation du ribosome ( fixation d’un facteur d’élongation) :GTP ------> GDP + Pi +E4.
5) Utilisation de GTP : GTP--------->GDP +Pi + E5
E1 et E5 sont négligeables par rapport à E2 et E3, en effet , ils comptent pour la synthèse de toute la protéine, E2 et E3 sont requis pur chaque ajout d’aa à la chaîne polypeptidique.
BILAN :pour chaque liaison amide, besoin de l’énergie de 4 liaisons riches en énergie : dont deux sont apportées par un ATP et deux autres par deux GTP.L’ancrage intime de ces facteurs de largage , a une influence sur la conformation de protéines ribosomiales et modifie l’action de la peptidyl transférase qui va hydrolyser la liaison peptidyl ARNt, corrélativement à l’hydrolyse d’un GTP en GDP.
C’est le prix à payer pour un fidélité de traduction optimum
3.4.6 Le « Wobble » ou base fluctuante
Le code génétique se compose de 61 codons qui ont un sens. Il faudrait donc s’attendre à trouver dans le cytoplasme 61 ARNt différents. D’après l’hypothèse du Wobble de Crick, 32 ARNt seraient suffisants pour véhiculer les 20 acides aminés différents.Un ARNt ne transporte qu’un seul acide aminé mais peu reconnaître plusieurs codons codant pour un même acide aminé .La base Wobble serait la première base de l’anticodon, correspondant à la troisième base du codon, il s’agit d’un appariement non classique.Ainsi pour l’Arginine, l’anticodon UCU peut reconnaître les codons AGA et AGG.En faisant le total de tous les Wobble, on arrive à un total de 32 ARNt et non 61, ce qui représente une économie pour la cellule. De plus, il semblerait que ces appariements atypiques sont plus lâches et favorisent une accélération de la traduction.
Il faut comprendre quand le ribosome se fixe sur l'ARNm pour le lire, on le divise en codons. Les ARNt (molécules qui apportent les AA au niveau du ribosome pour créer la protéine) ont un anticodon, c'est une séquence de 3 bases qui se fixe par complémentarité au codon c'est ainsi qu'un codon correspond à un AA. Mais la première base de l'anticodon qui se complémente avec la 3éme base du codon ne suit pas la complémentarité de Watson et Crick), c'est à dire qu'il peut se complémenter avec une autre base (ce qui explique la redondance du code génétique). Cette règle est la suivante :
G (première base de l'anticodon) peut reconnaître U/C (3ème base du codon)
I (première base de l'anticodon) peut reconnaître U/C/A (3ème base du codon)
I =Inosine=A désaminée
| Anticodon position 1 | Codon position 3 |
| I | U,C,A |
| U | A,G |
| G | C,U |
Différentes recherches son menées dans ce domaine, notamment la structure secondaire des ARNt. Ces recherches ont permis de déceler des appariement atypiques encore plus lâches que ceux du type Wobble. Rappelons qu’à priori, tous les appariements sont possibles.
Les différences d’énergie libre correspondant aux appariements peuvent être évaluées ( méthodes thermodynamiques, fonction ?
) , elles permettent de prévoir et d’expliquer la structure secondaire des ARNt et … leur structures tertiaires ( repliements de la molécule).
III.7.4.3Notion de séquence signal et modifications post traductionnelles des protéines
A) Généralités.
Les protéines synthétisées dans une celule peuvent:
- Rester dans le cytosol.
- Intégrer la membrane plasmique ou/et les membranes d’organites intra-cellulaires.
- Etre sécrétées à l’extérieur de la cellule.
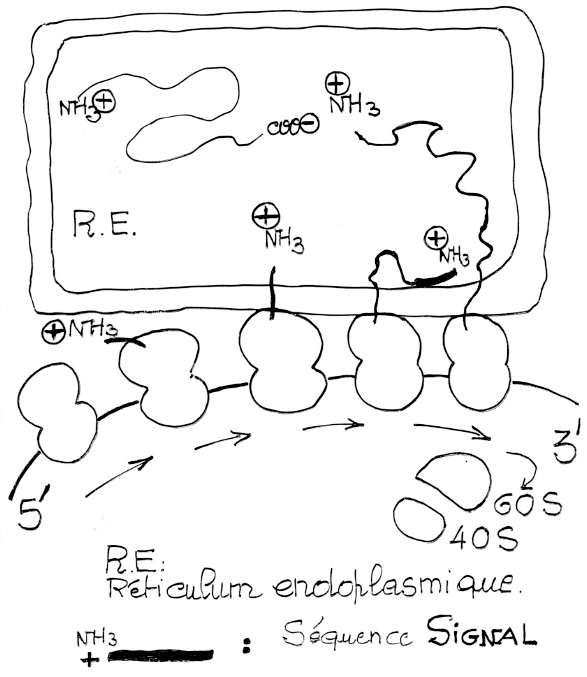
Une répartition s’effectue entre ces différentes destinations. Les protéines destinées à rester dans le cytosol sont synthétisées par des ribosomes non liés à une membrane cellulaire. Les protéines destinées à être sécrétées ou intégrées des membranes sont synthétisées par des ribosomes liés: soit à la membrane plasmique chez les procaryotes, soit à la membrane externe du réticulum endoplasmique chez les eucaryotes.
B Quelques précisions
Les protéines sécrétées ou membranaires possèdent à leur extrémité N-terminale une séquence courte d’acides aminés (une vingtaine en général) appelée séquence signal.
Cette séquence est particulièrement riche en acides aminés hydrophobes.
Chez les eucaryotes, au début de la synthèse de ce type de protéines, la chaîne polypeptidique est insérée dès le commencement de sa synthèse dans la citerne du réticulum endoplasmique. Cette opération complexe fait intervenir une reconnaissance spécifique de la séquence signal par des récepteurs situés sur la membrane externe du réticulum endoplasmique. Après cette rerconnaissance, la séquence signal est à l’intérieur de la citerne et la phase d’élongation de la synthèse protéique se poursuit. La séquence signal sera éliminée dans la protéine exportée de la cellule par un clivage protéolytique produit par une enzyme située à la face interne de la membrane du réticulum endoplasmique.
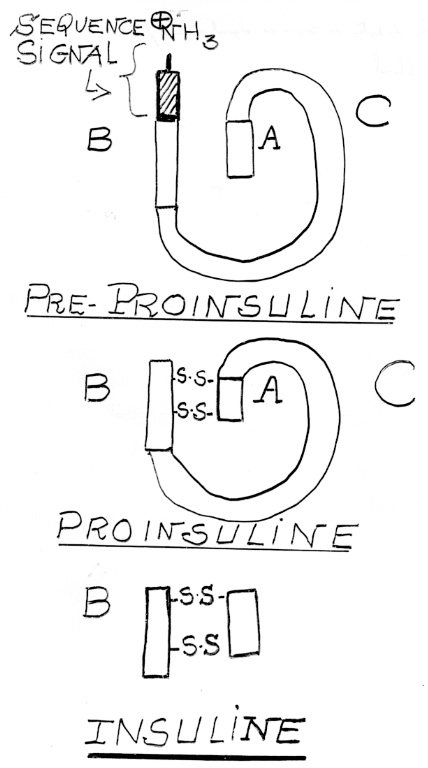
C. Exemple classique de protéine avec séquence signal, l’insuline
L’insuline est une hormone polypeptidique produite par les cellules de Langerhans du pancréas. Elle est synthétisée sous forme de pré-proinsuline avec une séquence signal à son extrémité N-terminale. Cette séquence signal est ensuite clivée dans le réticulum endoplasmique pour donner de la proinsuline. Un peptide appelé peptide C sera ensuite clivé dans les granules de sécrétion issus de l’appareil de Golgi et l’insuline active sera finalement produite sous forme de deux chaînes polypeptidiques (A et B) unies par des ponts disulfure (-S-S-).
D. Modifications post traductionnelles des protéines
(Rem : faire la liaison avec le contenu des notes de biologie cellulaire)
De nombreuses modifications sont possibles après la synthèse des protéines. Ces modifications peuvent être réversibles ou permanentes.
Modifications réversibles
Nous nous limiterons aux eucaryotes. Des groupements phosphates (phosphorylation) peuvent être ajoutés sur des résidus sérine, thréonine ou tyrosine. Des groupements acétyles (CH3-CO-) peuvent être ajoutés par exemple sur des résidus lysine.
Modifications permanentes
Ces modifications permanentes peuvent concerner:
La chaîne polypeptidique.
La chaîne polypeptidique peut être clivée à son extrémité N-terminale (qui comporte de la méthionine chez les eucaryotes). Ce résidu est en principe éliminé. Chez les procaryotes, l’extrémité N-terminale qui correspond à la formyl-méthionine est en principe seulement déformylée.
Les clivages par des enzymes de protéolyse des précurseurs de nombreuses protéines sécrétées rentrent également dans ce cadre.
Formation de ponts disulfure.
La formation de ponts disulfure entre deux résidus de cystéines est un événement majeur de la formation de la structure des protéines.
Hydroxylation des résidus proline et lysine dans le cadre du collagène
Addition de lipides.
Voir Biologie cellulaire, RE et Appareil de Golgi
Addition de sucres
La plupart des protéines sécrétées possèdent des structures oligosaccharidiques. Ce sont des glycoprotéines. L’addition des structures oligosaccharidiques peut se réaliser soit sur des résidus asparagine par des liaisons N-glycosidiques entre le -CO-NH2 de l’asparagine et un OH de la chaîne oligosaccharidique, c’est une N-glycosylation, soit plus rarement entre l’OH de la thréonine ou de la sérine et un OH de la chaîne oligosaccharidique c’est une O-glycosylation.
Date de dernière mise à jour : 22/03/2018
Ajouter un commentaire