- Accueil
- D.Sciences biologiques
- II.Biologie moléculaire COMPLET
- bio moléculaire 8
bio moléculaire 8
III.5 . Le contrôle de la transcription chez les Eucaryotes
III.5.1 Le complexe d'initiation.
A la différence de l’ARN polymérase des procaryotes, l’ARN polymérase II des eucaryotes ne se fixe pas directement sur le promoteur. Elle se fixe par intermédiaire de facteurs généraux de la transcription comprenant plusieurs protéines dénommées TF pour « transcription factor » et II pour l’ARN polymérase II: TFIIA,
TIIB.... Ces protéines associées à l’ARN polymérase II constituent le complexe d’initiation de la transcription et catalysent la formation de la première liaison phosphodiester entre les deux premiers nucléotides du mARN. Lorsque le site promoteur est libéré par la progression de l’ARN polymérase II sur l’ADN constituant la phase d’élongation, un autre complexe d’initiation peut se mettre en place.
III.5.1.1 Les facteurs généraux de la transcription et la formation du complexe basal d'initiation
Une succession d’étapes met en jeu des éléments du promoteur, l’ARN polymérase II et des facteurs protéiques généraux de la transcription.
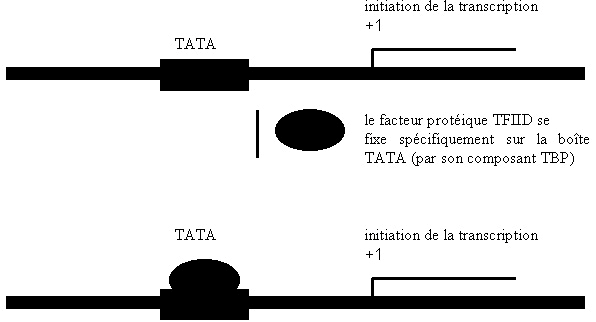
1/. La première étape est constituée par la fixation du facteur de transcription TFIID sur la boîte TATA. Cette fixation est realisée par l’intermédiaire d’un des composants du facteur TFIID qui est appelé TBP ( pour « TATA box binding protein »).
Un facteur additionnel TFIIA stabilise l’association facteur TFIID et boîte TATA. Il n’est pas représenté sur les schémas suivants. Le facteur TFIID comporte des facteurs appelés TAFII (« transcription activating factors ») Ces facteurs TAFII permettent l’interaction entre le facteur TFIIID et des éléments activateurs situés en amont de la boîte TATA.
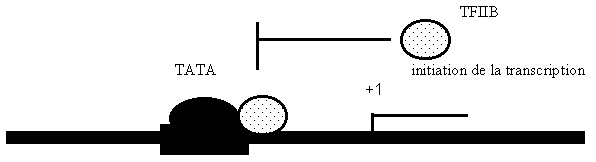
2/. Puis le facteur TFIIB se fixe sur le facteur TFIID fixé sur la boîte TATA.
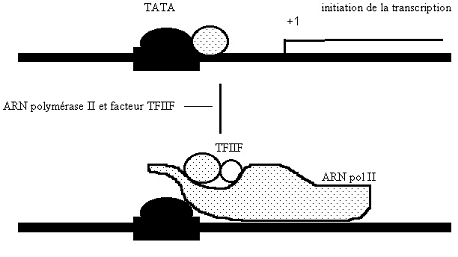
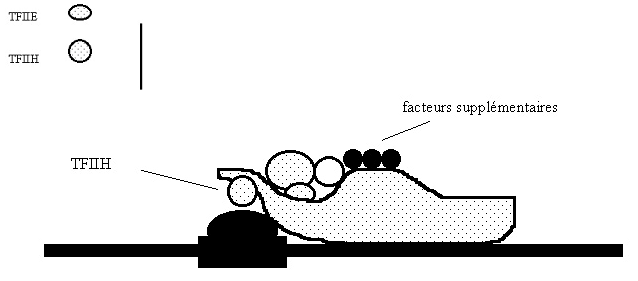
4/. Les facteurs TFIIE et TFII H se fixent, suivis par des facteurs supplémentaires complétant le complexe de transcription:
Le facteur TFIIH présente une activité protéine kinase. En présence d’ATP, une phosphorylation de l’ARN polymérase est réalisée sur la plus grosse sous-unité de l’enzyme riche en sérine et en thréonine (partie C-terminale).
5/. La phosphorylation sur sites spécifiques déclenche le début de la transcription:
De plus, arrivée au bout du promoteur, l’ARN polymérase II doit être libérée du complexe des facteurs de transcription généraux pour démarrer la transcription.
La phosphorylation d’une des sous-unités de l’ARN polymérase est indispensable pour déplacer l’enzyme du complexe d’initiation de la transcription.
III.5.2 Définition des facteurs CIS et TRANS régulateurs de la transcription
Il existe toute une série de séquences nucléotidiques comportant chacune un nombre limité de nucléotides (6-8 nucléotides le plus souvent) et dispersées généralement en 5’du gène, ces séquences sont appelées éléments cis-régulateurs. Ces séquences sont remarquablement conservées dans les
gènes de nombreuses espèces. Elles vont fixer des facteurs dits trans-régulateurs.
2.1. Facteurs cis-régulateurs de la transcription.
Quand un facteur trans-régulateur se fixe sur une séquence cis-régulatrice de l’ADN, la quantité de mARN synthétisé est brusquement modifiée, soit augmentée, soit diminuée. Les séquences activatrices (séquences « enhancer ») peuvent être situées à des distances très importantes du promoteur du gène (jusqu’à quelques dizaines de kilobases) et ceci en amont ou en aval du gène. Des séquences extinctrices (séquences « silencer ») ont un effet opposé aux séquences activatrices. Elles peuvent être également situées très à distance du promoteur du gène. C’est bien entendu la structure tridimensionnelle de l’ADN qui permet de rapprocher ces
éléments régulateurs du gène à transcrire.
Ainsi, pour nous limiter à quelques exemples, le facteur général de transcription TFIIID peut être considéré comme un facteur trans-régulateur. Il se fixe sur un élément cis-régulateur qui est la boîte TATA.
La boîte GC, élément cis-régulateur va fixer un facteur trans-régulateur, la protéine Sp1.
La boîte CCAAT, élément cis-régulateur va fixer le facteur CTF (« pour CCAAT binding transcription factor »).
2.2. Facteurs trans-régulateurs de la transcription.
Les facteurs trans-régulateurs sont des protéines particulières produites par d’autres gènes. Ces protéines présentent des caractéristiques structurales communes,
avec au moins au minimum deux domaines :
- Tout d’abord un domaine de fixation à l’ADN. Des motifs structuraux particuliers ont été décrits avec :
- Les doigts de zinc. Ce type de protéine comporte des éléments répétitifs avec une forme de doigt de gant. On peut retrouver de nombreux doigts. Ainsi, la protéine Sp1 qui se fixe sur les boîtes GC possèdent trois doigts de zinc. L’ion Zn2+ sert à stabiliser le motif sous forme de doigt.
- Les glissières à leucine. Les protéines avec glissière à leucine se fixent à l’ADN sous forme de dimères. Elles possèdent deux régions importantes. Une région à base du dimère, constituée par deux hélices alpha face à face riches en leucine et interagissant par des liaisons de type hydrophobe. Une autre région riche en
charges positives se fixe sur les groupes phosphates de l’ADN.
- Les homéodomaines à charges positives avec des structures hélice/boucle/hélice, hélice/tour/hélice (voir cours sur les protéines).
Un autre domaine, dit d’action sur la transcription. On peut rencontrer: différents types de domaines d’activation de la transcription. Des domaines riches en glutamine, des domaines riches en proline et des domaines organisés en hélice alpha riches en résidus à charge négative (hélice alpha-acide).
Certains facteurs trans-régulateurs possèdent un troisième domaine qui permet de fixer un élément annexe permettant de réaliser l’action d’un message extérieur à la cellule, comme un message hormonal. On a pu ainsi définir la superfamille des récepteurs nucléaires (stéroïdes,vitamines D, hormones thyroïdiennes, acides rétinoïques, acides gras polyinsaturés...).
Enfin, l’action des facteurs trans peut être elle-même régulée par d’autres facteurs avec modification des facteurs trans en réponse à une stimulation extérieure:
phosphorylation, protéolyse etc...
2.3. Approches intégrées.
Nous avons vu qu’il existe des séquences cis-régulatrices situées en 5’ du gène considéré. On les appelle des séquences régulatrices d’amont. Toute une série de séquences sur lesquels se fixent spécifiquement des facteurs transcriptionnels (ou facteurs trans) y sont retrouvées en plus des boîtes GC et CAAT. Au sens strict du mot promoteur d’un gène, celui-ci correspond à la région où se fixe l’ARN polymérase II (donc au niveau de la boîte TATA) jusqu’au site d’initiation de la transcription.
Certaines de ces séquences régulatrices d’amont confèrent une spécificité tissulaire d’expression aux gènes qui les possèdent. D’autres séquences sont la cible de
facteurs transcriptionnels dont la fixation ou l’activation sont sous le contrôle de stimulus extra- ou intra-cellulaires (hormones par exemple). Ces séquences cis de ce type sont appelées RE (réponse à un élément externe).
On peut donc dire que chaque gène est précédé par une série de séquences régulatrices d’amont séparées par des séquences non critiques sur l’activité transcriptionnelle. Le fait qu’un gène peut répondre ou non à un stimulus donné (par exemple: un choc thermique) dépend de la présence ou de l’absence de ces séquences. Ainsi, comme autre exemple, la présence d’une séquence ERE (réponse aux oestrogènes) en amont d’un gène donné permet à celui-ci d’être systématiquement activé, mais avec deux conditions supplémentaires: que la cellule possède des récepteurs pour les oestrogènes et que l’hormone soit présente.
2.4. Importance de la chromatine, méthylation des cytosines.
La chromatine est constitué d'un mélange d'ADN et de protéines. Il est important de rappeler que la chromatine est facilement observable après coloration dans les noyaux de cellules à l'interphase. Des nucléosomes forment un élément essentiel de la chromatine. Il sont constitués par environ 200 paires de bases d'ADN associées à un octamère d'histones. Chaque octamère comporte deux copies de chacune des histones : H2A,H2B, H3 et H4. Les histones H3 et H4 sont parmi les protéines les plus conservées sur le plan de leur séquence, ceci suggère que leurs fonctions sont identiques chez tous les eucaryotes. Ces histones constituent les histones de noyau. Par contre, l'hystone H1 bien que faisant partie du nucléosome peut être enlevée sans affecter la structure de celui-ci ce qui suggère sa localisation externe. Les hystones peuvent présenter des modifications transitoires sur certains groupement de leurs chaines polypeptidiques. Ainsi, des acétylations
ou des méthylations sont possibles et réversibles sur des résidus lysine. Des phosphorylations sont également réversibles sur les groupements OH,particulièrement au niveau de la sérine. Ces modifications sont transitoires et changent la charge de la molécule ainsi que ses intéractions avec les groupements phosphates de la molécule d'ADN. Enfin, des protéines non histones interagissent au niveau de la chromatine.
Deux états de la chromatine sont importants à connaitre et sont facilement mis en évidence par les colorations spécifiques
- l'hétérochromatine : ce mot désigne les région du génome qui sont en permanence dans un état hautement condensé. Ces régions ne sont pas transcrites et se répliquent tardivement. L'hétérochromatine peut être facultative ou constitutive. L'hétérochromatine constitutive reste constamment sous forme condensée et ne contient pas de gène fonctionels. L'hétérochromatine facultative peut se transformer en euchromatine.
- l'euchromatine est à l'opposé de l'hétérochromatine puisque les régions correspondantes sont activement transcrites et se répliquent précocément.
Quand la chromatine est digérée par l'enzyme DNase I, le premier effet est l'apparition de cassures en des sites spécifiques appelés sites hypersensibles. Ces sites représentent la disponibilité en ADN dans la chromatine. L'ADN à ce niveau n'est pas protégé par les octamères d'histones, bien que ceci n'impliquent pas nécessairement qu'il soit libre de protéines. Généralement, des gènes activement transcrits sont préférentiellement sensibles à l'enzyme DNase I. On peut également dire que cette sensibilité est une caractéristique des gènes qui sont capables d'être transcrits.
Il a été démontré chez les eucaryotes que les dinucléotides CG (ou CpG, p pour phosphate) sont souvent méthylés au niveau de la cytosine. Les génomes chez les eucaryotes ne sont pas méthylés de manière uniforme. Approximativement, 70 à 80% des sites CpG contiennent des méthyl-cytosines chez la plupart des vertébrés y compris chez l’Homme. Les méthylations de l’ADN concernent les régions régulatrices en 5’ des gènes. Environ la moitié des gènes chez la souris et chez l’Homme contiennent des ilôts CpG. Ce sont principalement les gènes domestiques (ou« housekeeping genes »). gènes spécifiant des protéines nécessaires à des fonctions communes à toutes les cellules, et dont l’expression est par conséquent ubiquitaire (par exemple, les enzymes de la glycolyse) qui en contiennent. Cependant, ces méthylations concernent également les gènes avec une expression tissulaire restreinte.
Les enzymes qui transfèrent des groupes méthyles à la cytosine en position 5 sur le cycle sont des cytosine 5-méthyltransférases ou DNA méthyltransérases. L’activité transcriptionnelle de nombreux gènes est étroitement dépendante de la méthylation de certaines zones de régulation de ceux-ci. Plusieurs hypothèses
ont été formulées pour expliquer la répression de l’activité transcriptionnelle par la méthylation de l’ADN. Il a été évoqué la possibilité d’une diminution
des interactions entre certains facteurs transcriptionnels et leurs sites spécifiques après méthylation de ces derniers. Il a été également suggéré que des liaisons directes entre des répresseurs transcriptionnels spécifiques et des sites ADN méthylés diminuerait l’activité transcriptionnelle. Enfin, plus récemment, la modification de la structure chromatinienne par la méthylation de l’ADN a été proposé.
Seulement 1 à 2 % de l'ADN code pour des protéines
III.6 Rôle des introns
III.6.1 Fonction catalytique
Rappelons qu’ils ont été découverts chez les eucaryotes supérieurs.Certains procaryotes possèdent des introns dans leurs gènes (archébactéries).Le rôle des introns est encore discuté. Cependant, quelques idées semblent émerger. Les introns pourraient faciliter l’élaboration de gènes complexes en délimitant des séquences codantes et permettant ainsi la mobilité des exons dans le génome. Enfin, l’absence d’introns dans un génome bactérien pourrait signifier que la bactérie est parvenue au stade ultime de son évolution et qu’elle aurait perdu toute possibilité d’évoluer vers la complexité.
On a également découvert que certains ARN pouvait avoir une activité catalytique, ils sont appelés Ribosymes.
En fait l'ARN possède beaucoup d'atouts lui permettant d'avoir une activité catalytique appréciable, atouts que l'on retrouve pour la plupart dans les protéines :
- Structuration en domaines
- Repliement tridimensionnel complexe
- Capacité à fixer des ligands variés ( Aptamères )
- Affinité particulière pour les analogues d'états de transition
- Fixation spécifique d'ions métalliques
Le terme ribozyme désigne donc les ARNs possédant une activité enzymatique,bien que beaucoup de ribozymes naturels ne catalysent qu'une fois leur réaction
spécifique et donc ne sont pas en tant que tel des vraies enzymes. Les ribozymes naturels ne sont pas très nombreux et se cantonnent généralement à des
réactions de clivage / ligation sur les acides nucléiques. Néanmoins ils fournissent la base de travail pour l'élaboration de ribozymes artificiels in vitro qui, s'ils n'atteignent pas une complexité structurale comparable à celle des ribozymes naturels, et donc n'atteignent pas non plus leur efficacité, permettent d'élargir le répertoire des activités catalytiques des ribozymes
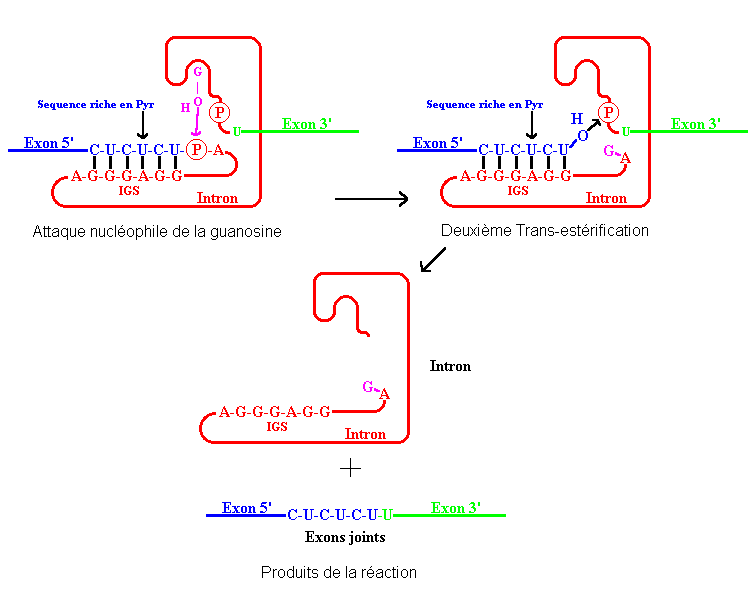
Les introns de groupe 1 ont été les premiers ribozymes a être découverts, par Tom Cech, dans le précurseur de l'ARNr 26S du protozoaire Tetrahymena thermophilus. Ils
ont la capacité d'autoépissage sans aucun autre apport exterieur que celui guanosine, phosphatée ou non, et de l'ion magnésium. Cet épissage est réalisé par 2 réactions de trans-estérification après l'attaque initiale de la guanosine sur le site d'épissage 5'.
Schéma récapitulatif des étapes de l'autoépissage
des introns de groupe I
Les différents introns de groupe I, bien qu'ayant peu de similarité dans leur séquence, ont en commun de éléments courts de structure primaire. Ces éléments
appartiennent à des domaines indispensables pour le repliement correct de l'ensemble, amenant à la structure catalytiquement active. Le site actif de la
catalyse nait ici principalement de 3 éléments :
o Le premier élément important est la création d'une poche de fixation pour la guanosine, qui peut discriminer entre la guanosine et les autres nucléosides et qui la
positionne optimalement pour une attaque nucléophile sur le site d'épissage en 5'.
o L'alignement du site d'épissage est réalisé par l'appariement d'une séquence riche en pyrimidine de l'exon en 5' avec une séquence guide (IGS pour Internal Guide Sequence) de l'intron.
o Le site d'épissage en 3', bien qu'éloigné dans la séquence, est aussi présent dans le site catalytique pour permettre lors de la 2ème transesterification la ligation
des 2 exons et la liberation de l'intron.
III.6.2 Evolution des organismes
Les premiers êtres unicellulaires possédaient un courant d’information cellulaire qui manquait de précision, d’où la nécessité de répéter plusieurs fois
un même gène codant pour de petites protéines.
Par la suite, un rapprochement de ces gènes par excision épissage a permis de réaliser des protéines plus grosses aux fonctions nouvelles .
III.6.3 Rôle dans l’excision épissage.
On remarque lors d’expériences de fusion de différentes souches de levure qu’une mutation au niveau de l’intron peut bloquer la procédure d’excision épissage d’un gène.
Importance des gènes en morceau dans la physiologie des organismes
Rappel : dans un organisme, toutes les cellules portent les mêmes gènes.
1er exemple : Cellules des CUB et de l’hypothalamus
Dans ces deux types de cellules, un gène à 6 exons va être transcrit en un même ARNpm.
Dans les cellules des CUB :
L’ARNm se composera des quatre premiers transcrits d’exons plus la séquence poly A. La traduction donnera le calcitonine, hormone impliquée dans
le métabolisme phosphocalcique de l’organisme .
Dans les cellules de l’hypothalamus :
L’ARNm se composera des transcrit des exons 1,2,3,5 et 6 plus séquence poly A.La traduction donnera une autre hormone C.G.R.P ( calcitonine gene related peptide) qui joue un rôle important en physiologie cardio-vasculaire.
Conclusion : Le choix des sites de coupure dépend des types cellulaires
Source de grande diversité avec une grande économie de moyens
Un gène peut coder pour plusieurs protéines,augmentation de la capacité de codage de l’ADN.
Remarque :
Cette propriété ( épissage alternatif) est à l’origine, en partie , de la diversité des Anticorps circulants.
Date de dernière mise à jour : 22/03/2018
Commentaires
-

1 ludi Le 01/05/2015
Au top le cours
-

2 NANCY Le 02/04/2013
SUPER -

3 bazile Le 02/04/2013
super les cours -

4 bazile Le 02/04/2013
c super ces cours
Ajouter un commentaire