- Accueil
- D.Sciences biologiques
- II.Biologie moléculaire COMPLET
- bio moléculaire 7
bio moléculaire 7
2 ème étape : élongation
- Formation de liaisons ester entre nucléotides, l’énergie de liaison est apportée par les nucléotides eux-mêmes.
- Progression de la transcription 5’ 3’
L’ARN polymérase peut être comparée à un curseur se déplaçant sur l’ADN
Après son passage, il y a restauration de ces liaisons.
Etape 3 : fin de la transcription
- Chez les procaryotes : Signaux de fin de transcription
1° Région à symétrie imparfaite ou palindrome imparfait
2° Région riche en A-T, région plus lâche, permettant à l’ARN polymérase de sortir de transcription.
5’ GCCGCCAC TTCCG CTGGCGGC ATTT 3’
3’ CGGCGGTG AAGGC GACCGCCG TAAA 5’
De plus au niveau du palindrome, se forme une boucle en épingla à cheveux ( self complémentarité ) ; elle sera responsable de l’arrêt de la transcription ( plus que la séquence riche en AT).
- Chez les eucaryotes
Le palindrome imparfait se trouve plus en amont
Séquence de fin de gène et( non de transcription ) : AATAAA, cette séquence est lue sur le brin d’ADN non transcrit.
L’ARN polymérase reconnaît ce signal, mais continuera à transcrire au delà.
Les transcrits seront raccourcis par la suite, ils se termineront par la séquence AAUAAA suivie de 10 à 15 nucléotides.
Remarques sur la transcription
- Choix du brin d’ADN copié par l’ARNase ?
C’est la direction de l’ARNase le long de l’ADN qui détermine le choix.
- Amplification des informations contenues dans l’ADN
Pour l’ARNm : 1 gène x ARNm y protéines.
X et y = +/- 1000
Pour ARNr et ARNr : plus de traduction
Conséquence : plusieurs copies identiques d’un même gène.
Les modifications post transcriptionnelles
------------------------------------------------------------------------------------------------------------------------------------------------------------
PREALABLES A L'ELONGATION
I. LE MECANISME GENERAL
La transcription est une biosynthèse d'ARN, elle ressemble par certains points au mécanisme de la réplication notamment en répondant au principe de complémentarité des bases mais comporte aussi des différences fondamentales avec celle-ci :
- Le processus de réplication, à chacun de ses cycles concerne la totalité du génome, ce n'est pas le cas de la transcription qui n'est pas fixe , seules de petites portions du génome sont copiées en ARN à une époque donnée de la vie de la cellule, et ce, en fonction du stade de développement, de l'environnement, ...
La transcription commence en un point précis de l'ADN génomial et se termine en un point également précis. L'espace entre ces deux points fixes est appelé unité de transcription.
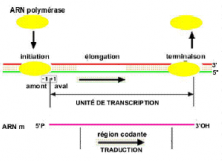
- Un seul brin de l'ADN est transcrit, c'est à dire sert de modèle à la polymérisation des ribonucléotides, en effet, en un endroit donné, un seul brin a un sens en terme de protéine. C'est pourquoi l'on écrit généralement une séquence d'ADN sous forme d'une succession de bases de 5' à 3', dans cette convention, l'on représente effectivement le brin qui possède le code ( brin codant), mais c'est l'autre brin ( 3' 5') dans cette convention qui est transcrit en ARN. Le résultat est une molécule d'ARN dont l'orientation 5' 3' correspond à l'orientation NH2--COOH de la protéine. La lecture du code se fait dans le même sens ( la traduction) se fait dans le même sens que la transcription.
- Remarquons que tout le produit de la transcription ne correspond pas à la séquence qui code de la protéine, côté 5' (amont) une séquence guide ou " leader" permet la fixation de l'ARN messager au ribosome.
La transcription est assurée par une ARN polymérase qui utilise l'ADN simple brin ( comme pour la réplication, une dénaturation locale de la molécule d'ADN est nécessaire) mais elle polymérise des ribonucléotide en regard de désoxyribonucléotides. Contrairement à l'ADN polymérase qui ne fait qu'allonger des séquences déjà existante, l'ARN polymérase peut réaliser un dinucléotide au point d'initiation de la transcription.
De nombreux facteurs protéiques interviennent également pour assurer l'initiation, l'élongation, la terminaison, mais ce ne sont pas les mêmes que ceux qui jouent ce rôle dans la réplication. Il existe des différences sensibles entre la transcription chez les procaryotes et celle des eucaryotes et des différences encore plus importante dans ses mécanismes de contrôles. C'est pourquoi leur étude sera séparée
I.I Transcription chez les procaryotes .
L'ARN polymérase d'E.coli est une enzyme assez complexe composée de quatre sous-unités , deux alpha, une bêta et une bêta ' et d'autres éléments protéiques qui établissent des liaisons temporaires avec ce complexe.. Cet enzyme réalise la polymérisation des ribonucléotides face aux désoxyribonucléotides d'un brin d'ADN mais in vitro commence n'importe où et s'arrête n'importe où. Une protéine particulière, le facteur sigma, est capable, in vivo, de reconnaître des séquences spécifiques situées légèrement en amont du point d'initiation qui constituent le promoteur . L'installation de l'ARN polymérase au niveau précis de ce promoteur (grâce au facteur alpha) va permettre la transcription du bon nucléotide au niveau du bon bout. C'est le point crucial de l'initiation. Au démarrage de l'élongation, le facteur sigma quitte le complexe. Cette étape marque le passage à l'allongement de la chaîne d'ARN dans le sens 5' 3' jusqu'au site de terminaison.
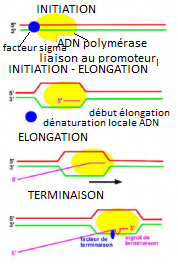
On voit en rose sur le dernier schéma : signal de terminaison . ( la figure est à remplacer ) et en bleu : facteur de terminaison .
Les schémas, ci-dessus impliquent que la transcription correcte, dans l'espace, d'une molécule d'ARN représentative d'une unité d'information repose sur le point d'initiation et sur le point de terminaison . comment ces nucléotides précis sont-ils déterminés ? ce sont les séquences promotrices et les sites de terminaison qui contiennent les informations nécessaires. Plus d'une centaine de promoteurs ont été séquencés et l'alignement des séquences par rapport au point de départ de la transcription fait ressortir ce que l'on appelle des séquences consensus , c'est à dire qui se retrouvent très fréquemment au même endroit dans différents promoteurs.

séquence consensus : TATAAT
Une première en position -10 est appelée boîte -10 ou boîte TATA ( TATA box) en raison de sa composition Une autre est une boîte -35 dont le consensus est TTGACA. Toute mutation dans cette région affecte l'initiation de la transcription soit dans le choix du brin à transcrire soit dans la position du point de départ. Des délétions qui modifient les distances entre ces séquences ou par rapport au point de départ affectent également la transcription.
Le promoteur est donc défini par des séquences d'initiation types en positions précises.
La terminaison semble pouvoir se faire selon au moins deux mécanismes, selon les renseignements apportés par les expériences de transcription in vitro, pour certains gènes, elle dépend de la structure secondaire prise par l'ARN lui-même. La structure de l'extrémité 3' de nombreux messagers se rapproche de celle présentée ici.  le signal de terminaison est ici reconnu après avoir été transcrit. La séquence polyuridilique en 3' implique une série de paires A-U à deux liaisons hydrogène seulement qui facilite la libération de la molécule d'ARN. dans d'autres cas, un facteur protéique spécifique, ce facteur est nécessaire pour la terminaison et la libération de la molécule d'ARN.
le signal de terminaison est ici reconnu après avoir été transcrit. La séquence polyuridilique en 3' implique une série de paires A-U à deux liaisons hydrogène seulement qui facilite la libération de la molécule d'ARN. dans d'autres cas, un facteur protéique spécifique, ce facteur est nécessaire pour la terminaison et la libération de la molécule d'ARN.
Remarquons que les trois grandes classes d'ARN : ARN messager, ARN de transfert, ARN ribosomial, sont synchronisés selon ce mécanisme de transcription. Il faut savoir que sur le plan qualitatif, les cistrons ( Un cistron est une région du génome correspondant à l'unité codant ou non une chaîne polypeptidique. Ce terme est synonyme de gène si ce gène code une protéine, le cistron comprend alors l'ensemble des codons qui code une protéine donnée) ribosomiques et de transfert sont peu diversifiés et occupent une partie petite du génome bien que sur le plan quantitatif leur produits semblent composer plus de 50% de l'ARN et induire ainsi que l'ARN messager peu représenté dans le cytoplasme peut l'être à partir de gènes très variés qui se répartissent sur une grande partie du génome. Ce paradoxe est accentué dans le cellule eucaryote.
I.2 Transcription chez les eucaryotes
Le principe est bien entendu le même,il s'agit de transcrire un code contenu dans la molécule d'ADN, mais les modalités sont différentes.
I.2.1 Les ARN polymérases
Alors qu'une seule enzyme synthétise l'ensemble des catégories d'ARN des procaryotes, dans la cellule eucaryote, trois enzymes ARN polymérases sont mises en jeu dans la transcription d'ensembles de cistrons différents.
- L'ARN polymérase A (ou I) transcrit les gènes ribosomiques ( qui restent souvent appelés cistrons ribosomiques). Elle assure la synthèse des ARN des ribosomes. Son lieu d'action est le nucléole . En effet, les très nombreux cistrons ribosomiques répétés ( c'est une de leurs caractéristiques ) sont groupés au niveau de loci précis : les organisateurs nucléolaires . A l'interphase, la transcription active de ces cistrons .se manifeste sous forme de structure cytologiques : le nucléole.
- L'ARN polymérase B ( ou II) réalise la synthèse de tous les ARN messagers nucléaires qui seront traduits en protéines. Ces gènes seront regroupés dans ce que l'on appelle les " gènes de la classe II).
- L'ARN polymérase C (ou III) assure la synthèse des petits ARN nucléaires ( ARN de transfert et ARN ribosomique 5S) et la transcription des gènes cytoplasmiques ( contenus dans les mitochondries et les plastes des végétaux).
Toutes ces polymérases sont des édifices protéiques complexes composées de quatres sous-unités principales rappelant celle des procaryotes. auxquelles s'ajoutent jusqu'à une douzaine de protomères. De plus ces complexes ne fonctionnent in vivo que grâce à l'intervention de nombreux autres facteurs protéiques établissant des relations temporaires avec le complexe enzymatique de base.
Ce que l'on connaît de la transcription des cistrons ribosomiques rappelle la transcription de l'ARN procaryotique ( ce qui ne veut pas dire que les systèmes de régulation soient identiques) et nous ne nous y attarderons pas.
I.2.2 Transcription des gènes exprimés en protéines.
La transcription des gènes de la classe II est assurée par l'ARN polymérase B , mais parmi eux il faut distinguer différents types d'organisation. Quelques rares gènes présentent des unités de transcription semblables à celles des procaryotes . Le plus souvent les gènes sont organisés en structures dites mosaïques de positions codantes ( les exons ) et de positions non codantes ( les introns ).
L'organisation d'un messager eucaryotique est présenté ci-dessous
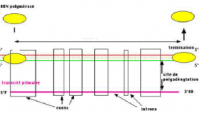
figure à remplacer
Etape 3 : fin de la transcription
- Chez les procaryotes : Signaux de fin de transcription
1° Région à symétrie imparfaite ou palindrome imparfait
2° Région riche en A-T, région plus lâche, permettant à l’ARN polymérase de sortir de transcription.
5’ GCCGCCAC TTCCG CTGGCGGC ATTT 3’ (ARN AUUU)
3’ CGGCGGTG AAGGC GACCGCCG TAAA 5’ ( ARN UAAA)
De plus au niveau du palindrome, se forme une boucle en épingla à cheveux ( self complémentarité ) ; elle sera responsable de l’arrêt de la transcription ( plus que la séquence riche en AT).
- Chez les eucaryotes
Le palindrome imparfait se trouve plus en amont
Séquence de fin de gène et( non de transcription ) : AATAAA, cette séquence est lue sur le brin d’ADN non transcrit.
L’ARN polymérase reconnaît ce signal, mais continuera à transcrire au delà.
Les transcrits seront raccourcis par la suite, ils se termineront par la séquence AAUAAA suivie de 10 à 15 nucléotides.
Remarques sur la transcription
- Choix du brin d’ADN copié par l’ARNase ?
C’est la direction de l’ARNase le long de l’ADN qui détermine le choix.
- Amplification des informations contenues dans l’ADN
Pour l’ARNm : 1 gène x ARNm y protéines.
X et y = +/- 1000
Pour ARNr et ARNr : plus de traduction
Conséquence : plusieurs copies identiques d’un même gène.
Les modifications post transcriptionnelles
Chez les procaryotes
ARNr : Précurseur qui sera ensuite clivé pour donner un ARNr stable.
ARNt : Précurseur qui subira :
- Des clivages
- Des additions ( CCA en 3’)
- Des modifications de bases ( bases atypiques).
ARNm : Pas de modifications. La traduction commence avant la fin de la transcription.
- Chez les eucaryotes.
a) Structure de l’ADN des eucaryotes par rapport à celui des procaryotes
L’ ADN d’un gène d’organisme eucaryote se subdivise en séquences appelées « exons » et « introns » qui alternent .
La transcription de ce type de gène appelé gène mosaïque ou en morceaux portera le nom de ARN pré messager ( ARNpm ) ou transcrit primaire.
Ce gène comprend en plus les régions 5’UTR et 3’UTR qui ne sont pas traduites. Par la suite, par un processus d’excision-épissage, ce transcrit donnera l’ARN messager qui ne porte plus que les parties correspondant aux exons ; l’information portée par les introns ne sera pas traduite.
b) Modifications subies par le transcrit primaire
1° Addition du cap à l’extrémité 5’
Le premier nucléotide du transcrit primaire commence par un groupement triphosphate.
La base correspondante est en principe une base purique (A ou G).
L'extrémité 5’ initiale du transcrit primaire peut alors être représentée par : 5’ pppA/GpNpNpNpN... avec p=groupement phosphate et N=A,T,C ou G.
En présence de GTP et d'une guanylyl transférase, on a la réaction suivante :
Gppp+pppApNpNp...----->GpppApNpNp...+pp+p
Le premier phosphate situé à l’extrémité 5’ du transcrit primaire va être éliminé au cours d’une soudure (par une liaison anhydride d’acide) avec du GMP (guanosine
monosphosphate) provenant du GTP. Il y a donc constitution d’une liaison inhabituelle: 5’à 5’ triphosphate. On parle de coiffe ou « cap ».
Cette coiffe en 5’ du transcrit primaire se met en place dès le début de la transcription.
Au niveau de la base de la guanosine terminale s’ajoute un groupement méthyle sur l’atome d’azote en 7. D’autres modifications peuvent se produire comme des méthylations en 2’ sur les riboses situés sur les deux premiers nucléotides de l’extrémité 5’ du transcrit primaire.
Il n’y aura donc plus de 5’ phosphate libre mais un OH libre en 3’ aux deux extrémités
La coiffe est indispensable pour :
- Protéger les mARN de l’attaque par des enzymes de dégradation ( phosphatases,polynucléases).
- Permettre la traduction, pas de cap chez les autres ARN, pas de traduction.
------------------------------------------------------------------------------------------------------------------------------------
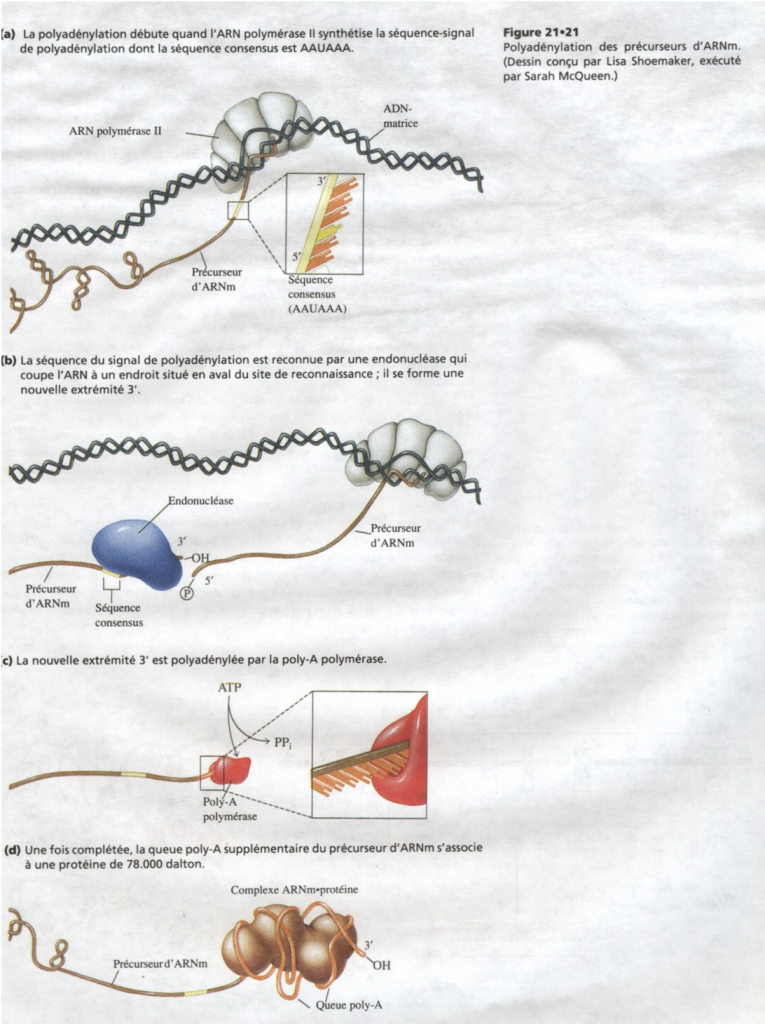
2° Addition de poly A à l’extrémité 3’
Addition de plus ou moins 100 nucléotides à Adénine à l’extrémité 3’.Dés que l’ADN polymérase II a transcrit au delà du bout 3’ de la séquence codante de l’ADN , l’ARN naissant est scindé au voisinage d’un site particulier dont la séquence consensus est AAUAAA : ce site est le site de polyadénylation.La scission se produit en
général à une distance de plus ou moins 10 à 20 nucléotides en aval de ce signal et dépend probablement d’autres séquences ainsi qu’éventuellement de la
structure du précurseur du ARNpm.La nouvelle extrémité 3’ créée ainsi sert d’amorce à l’addition récurrente d’ une série de résidus adénosine catalysée par la poly A polymérase.Dans cette réaction utilisant l’ATP, l’enzyme peut ajouter jusqu'à 250 nucléotides pour constituer un appendice de poly adénylate connu sous le nom de poly A qui s’associe à une protéine de 78.000 Daltons.
Les rôles de cette queue poly A seraient de :
- Permettre le passage de l’ARNm du noyau vers le cytoplasme
- Protéger l’ARNm au cours de la traduction.
3° Maturation du précurseur

Dans le noyau : excision des transcrit d’introns et épissage des transcrits d’exons.
Les enzymes d’excision épissage ne sont pas spécifiques du transcrit d’intron, elles reconnaissent la jonction transcrit d’intron – transcrit d’exon.
Ce mécanisme se déroule dans le noyau des eucaryotes. Chez les eucaryotes supérieurs, il a été démontré très tôt que l'ARN nucléaire était hétérogène et instable. Cet ARN a été appelé ARN nucléaire hétérogène (hnRNA). La forme physique du hnRNA est celle d'une particule ribonucléoprotéique dans laquelle le hnRNA est lié à des protéines.
Le transcrit primaire ou pré-mARN est inclus dans l'hnRNA, qui contient bien entendu d'autres transcrits.
Importance de la conservation de séquences situées à la jonction exon-intron.
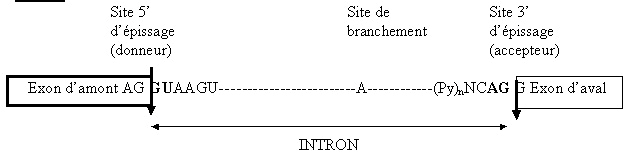
Au niveau du transcrit primaire: La séquence des bases d’un intron commence par GU et se termine par AG. La séquence consensus à l’extrémité 5’ des introns des vertébrés est GUAAGU ou GUGAGU. A l’extrémité 3’, la séquence consensus est un brin de dix pyrimidines (Py = U ou C) suivie par n’importe quelle base N (A, U, G ou C) puis par C (ou U) et se termine par la séquence invariante AG. L’exon d’amont se termine par CAG ou AAG. L’exon d’aval commence par une base G ou A. Le site de branchement (A) est localisé entre 20 et 50 nucléotides en amont du site 3’ d’épissage.
Le mécanisme de l’excision-épissage fait intervenir deux réactions de transestérification.
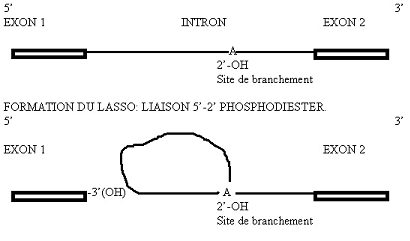
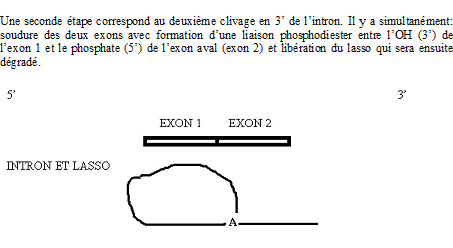
Dans la première étape, il y a formation d’un premier clivage en 5’ de l’intron. L’OH situé en 2’ du ribose appartenant à l’A du branchement vient se
souder à l’extrémité 5’-phosphate de l’intron. Il y a formation d’une liaison 5’-2’ phosphodiester. Il y a constitution d’une boucle ou lasso (« lariat »). L’extrémité 3’ de l’exon situé en amont du clivage (exon 1) présente un OH (3’) libre:
Ce mécanisme doit être tout à fait précis car le moindre décalage d’un nucléotide modifie le cadre de lecture lors de la traduction : protéine fortement modifiée.
Mécanisme chimique
VI.5 . Le contrôle de la transcription chez les Eucaryotes
Date de dernière mise à jour : 22/03/2018
Commentaires
-

1 Kasey Le 25/03/2017
you are in reality a just right webmaster. The web site loading speed is incredible. It sort of feels that you're doing any unique trick. In addition, The contents are masterwork. you have performed a great activity on this topic!
Ajouter un commentaire