- Accueil
- D.Sciences biologiques
- II.Biologie moléculaire COMPLET
- bio moléculaire 6
bio moléculaire 6
L’acide désoxyribonucléique (ADN ou DNA) est constitué de deux chaînes de nucléosides monophosphates liés chacun par une liaison ester entre son carbone 3’ (alcool secondaire) et le carbone 5’ (alcool primaire) du nucléotide suivant.
• Ces deux chaînes de nucléotides sont unies entre elles par des liaisons hydrogènes pour former un hybride en forme de double hélice (modèle de J.D. Watson et F.H.C. Crick, 1953) dont les
deux brins sont :
— antiparallèles : l’un est constitué d’un enchaînement commençant à gauche et se poursuivant vers la droite, l’autre commençant à droite et se poursuivant vers la gauche ;
— complémentaires : chaque adénine (A) d’un des deux brins est liée par deux liaisons hydrogène avec une thymine (T) de l’autre brin, et chaque guanine (G) d’un brin est liée par trois liaisons hydrogène avec une cytosine (C) de l’autre brin.
• De sorte que si on désigne les nucléotides par les lettres A, C, G et T en fonction des bases azotées qu’ils contiennent, on peut lire sur cette figure le texte suivant :
...AGAGTCGTCTCGAGTCA...
...TCTCAGCAGAGCTCAGT...
Séquence d'un gène Apo A- II
Ecrire à la suite les lettres A, C, G et T dans l’ordre où on rencontre les nucléotides sur l’un des brins du DNA de l’extrémité 5’ à l’extrémité 3’ aboutit à un texte indéchiffrable
• Pourtant le texte ainsi transcrit contient toute l’information nécessaire pour la synthèse d’une protéine (l’apolipoprotéine A-II). C’est pourquoi on appelle ce brin de DNA le brin « sens ». L’autre brin est le brin complémentaire ou brin « antisens ».
• L’ensemble de l’information génétique transmise par chacun des parents à un enfant (génome haploïde) peut s’écrire ainsi en 3 milliards de lettres (une bibliothèque de 7000 livres de 300 pages chacun !)
• Les protéines du noyau savent chercher sur les brins de la double hélice du DNA des suites de lettres (séquences) sur lesquelles elles se fixent spécifiquement. Les effets de cette fixationde protéines sur le DNA vont permettre l’expression de l’information contenue dans le DNA pour faire vivre un individu.
Pour permettre la biosynthèse d’une protéine, il doit y avoir dans la structure du DNA d’un sujet, une séquence de nucléotides qui constitue le gène de cette protéine.
• Dans la séquence du gène on distingue des séquences en amont (éléments régulateurs, promoteur),un site d’initiation de la transcription, une suite variable d’exons et d’introns : exons non-traduits ou traduits, introns comportant éventuellement d’autres séquences régulatrices,et enfin la fin de la transcription à la fin du dernier exon.
• L’ensemble des mécanismes qui à partir de la séquence du gène conduisent à la productiond’un acide ribonucléique ou d’une protéine est désigné sous le terme d’expression de ce gène.
L’expression d’un gène est une suite de synthèses chimiques et de réactions aboutissant à la production d’un acide ribonucléique ou d’une protéine.
• Dans un premier temps a lieu la synthèse d’un acide ribonucléique, dont la séquence est complémentaire d’un des deux brins du gène donc identique à celle de l’autre brin : c’est la transcription.
• La séquence de l’acide ribonucléique est identique à celle du brin sens et orientée dans lemême sens. Elle se construit de 5’ vers 3’ en complémentarité de celle qui est « lue » sur le brin antisens.
• Après des réactions de maturation le transcrit primaire (RNA) devient RNA messager (mRNA)et sort du noyau vers le cytoplasme.
• Dans le second temps, le RNA messager est « lu » par groupe de trois nucléotides (lettres) grâce à un RNA complémentaire qui porte l’acide aminé correspondant.
• La « lecture » du mRNA se fait de 5’ vers 3’ et la synthèse de la protéine se fait en même temps de l’extrémité NH2 terminale vers l’extrémité COOH terminale.
• Après des réactions de maturation, la protéine « mature » est prête à remplir sa fonction dans la cellule.
VI.LA TRANSCRIPTION .
VI.1Définition
Il s’agit d’un mécanisme de synthèse d’ARN à partir d’un fragment d’ADN appelé gène. Dans cette procédure, un seul brin du fragment d’ADN concerné est copié.
ou
VI.2 Caractéristiques de la transcription
La synthèse s’opère dans le sens 5’ 3’, de façon antiparallèle et complémentaire .
VI.3 Eléments nécessaires à la transcription
La transcription nécessite :
- Des ribonucléotides activés : ATP , GTP , CTP , UTP.
- Une enzyme : l’ARN polymérase, qui catalysera la formation de la chaîne polynucléotidique. Les sels de Magnésium sont indispensables au fonctionnement de cette enzyme.
La transcription fait donc intervenir une activité enzymatique nommée ARN polymérase holoenzyme. Cet énorme complexe enzymatique déroule et disjoint les deux brins de l'ADN hélicoïdale. Il recrute les mononucléotides du futur brin d'ARN et les assemble par complémentarité avec les bases de la séquence du brin d'ADN.
- Un modèle d’ADN.
VI.4 Différentes étapes de la transcription
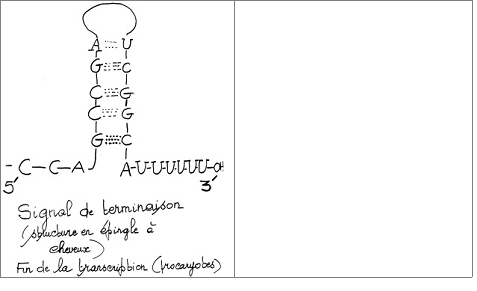
La transcription porte sur un gène bien défini , donc elle est sélective.
Comment les séquences à traduire sont-elles reconnue par l’ARN ase ?
1ère étape : début de la transcription ( initiation).
On donne par convention, le chiffre +1 au premier nucléotide à partir duquel la transcription commence. Le chiffre -1 désigne le nucléotide qui le précède.
Le signal pour initier une transcription correcte est appelé promoteur. Cette séquence d’ADN est située juste avant le début de la région où commencera la transcription.
Les promoteurs les plus simples sont les promoteurs des gènes des procaryotes. Nous étudierons pour commencer essentiellement ceux-ci, les promoteurs des gènes des eucaryotes étant beaucoup plus complexes.
Chez les procaryotes.
Les promoteurs sont pour la plupart situés en 5’ (ou en amont) du site +1 d’initiation de la transcription et sont constitués de plus ou moins 40 paires de nucléotides. Ces promoteurs ne sont donc pas transcrits.
La comparaison des séquences des promoteurs de nombreux gènes de procaryotes (E. coli) a montré la présence de séquences nucléotidiques courtes présentant une grande similitude de structure. Deux séquences nucléotidiques sont remarquables.
- Une séquence située entre -30 et -35 paires de bases environ en amont de l’origine de la transcription est appelée séquence -35. Cette séquence -35 présente un motif de six bases bien conservées: TTGACA.
<-- amont aval -->
5'-XXXXXXXPPPPPXXXXXXPPPPPPXXXXGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGXXXX-3'
-35 -10 +1 gène d'intérêt
TTGACA TATAAT
- Enfin, à environ 10 nucléotides en amont de l’origine de la transcription, on retrouve un motif TATAAT. Cette séquence dite séquence -10 est appelée également boîte de PRIBNOW.
La distance séparant les sites -35 et -10 est comprise entre 16 et 18 pb dans 90% des promoteurs des procaryotes. Le maintien d'une telle distance est d'importance pour la fixation de l'ARN polymérase.
Chez les procaryotes, les ARN présents: ARN messagers, ARN de transfert et ARN ribosomaux sont synthétisés par la même ARN-polymérase. L’ARN-polymérase d’E. coli est composée de 5 sous-unités qui sont des chaînes polypeptidiques distinctes, 2 chaînes alpha, 1 chaîne béta, 1 chaîne béta’ et 1 chaîne s (sigma). L’ensemble de ces cinq sous-unités constitue l’holoenzyme de l’ARN-polymérase. La sous-unité sigma n’est pas associée en permanence à ce complexe mais de manière transitoire. Sa présence est nécessaire pour permettre à l’ARN-polymérase de reconnaître les sites promoteurs. Après l’initiation, cette sous-unité sigma se dissocie. Le noyau de l’enzyme (« core ») est constitué par les sous-unités (alpha)2bétabéta’ et il contient le site catalytique de l’enzyme responsable de l’élongation de la chaîne polynucléotidique.
Le complexe formé par le noyau de l’enzyme et le facteur s vient se positionner sur le promoteur. Il recouvre une région située entre les positions -60 et +20. Dès lors, placée sur cette région, l’ARN polymérase protège l’ADN de l’action de désoxynucléases. Cet effet de protection a été démontré par des expériences de "foot-printing". Une dénaturation locale de l’ADN se fait de la position -10 à la position +1. Dès la formation de la première liaison phosphodiester du mARN, le facteur s est relâché. Des facteurs additionnels protéiques et le degré d’enroulement de l’ADN influent sur l’activité transcriptionnelle. Il est évident que la transcription a un effet très important sur la structure locale de l'ADN. Dès lors, l'action des enzymes suivantes : gyrase (qui introduit des super-tours négatifs) et topoisomérase I (qui enlève des super-tours négatifs) est essentielle en avant et en arrière de l'ARN polymérase au cours de sa progression le long de l'ADN.
Chez les Eucaryotes
1) Initiation
On connaît moins bien les signaux de début de transcription.
Contrairement aux procaryotes qui possèdent qu’une seule ARN polymérase, les eucaryotes ont trois ARN polymérases qui assurent la synthèse des différents ARN. La transcription implique donc:
1. L’ARN polymérase I. Elle transcrit les rARN: 5,8 S, 18 S et 28 S, sauf le rARN 5 S.
2. L’ARN polymérase II. Elle transcrit les précurseurs des mARN ou transcrits
3. L’ARN polymérase III. Elle transcrit les tARN, les rARN 5 S et une faible fraction des ARN nucléaires.
Ces trois ARN polymérases ont été initialement distinguées par leur sensibilité à un poison isolé des champignons (amanites phalloïdes): l’a-amanitine. Des trois ARN polymérases, l’ARN polymérase II est la seule très sensible à ce poison. Ces ARN polymérases sont localisées dans le noyau de la cellule.
La transcription par chacune des ces trois ARN-polymérases implique les trois même phases que chez les Procaryotes à savoir initiation , élongation, terminaison.
Les ARN ase III reconnaîtrait :
- Vers la séquence –30 une TATA box ( équivalent de la PRIBNOW box)
'-XXXXXXXXXXXXXXXXXXXXXPPPPXXXXXXXXXXXXXXXXXXXXXXXXXXGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGG-3'
-30 gène d'intérêt
TATA box
- Vers la séquence –130, une CAAT box
Si on provoque une mutation au niveau de la TATA box : 50 à 70% d’inhibition de la transcription.
La transcription par chacune des ces trois ARN-polymérases implique les trois étapes suivantes:
Toutes les ARN polymérases ont besoin de facteurs multiples pour se positionner au point de départ de la transcription. Il existe des facteurs généraux de la transcription qui contribuent à un niveau bas de transcription. Des facteurs additionnels par exemple activateurs peuvent augmenter la transcription au-delà du niveau basal.
En détail
L’expression du génome aboutit à la synthèse dans les cellules de macromolécules acides nucléiques et protéines, dont la structure primaire est déterminée par celle du DNA.
• Cette expression se fait par deux mécanismes principaux :
— la structure primaire du DNA s’exprime d’abord par la synthèse d’acides ribonucléiques dont la structure primaire est parallèle à celle du DNA. C’est la transcription.
— la structure transcrite sur certains RNA, dits « messagers », s’exprime enfin par la synthèse de protéines dont la structure primaire traduit en acides aminés l’information portée par la structure primaire du DNA. C’est la traduction.
• La transcription est conduite par plusieurs enzymes :
— RNA-polymérase I qui synthétise les RNA cytoplasmiques : RNA ribosomiques (18 S-5,8 S- 28 S)
— RNA-polymérase II qui synthétise les RNA messagers qui contiennent l’information destinée à la traduction et certains des snRNA
— RNA-polymérase III qui synthétise les petits RNA (tRNA, rRNA 5 S, snRNA, 7SLRNA).
Toutes les ARN polymérases des eucaryotes sont des protéines constituées de nombreuses sous-unités (typiquement de 8 à 14). La plus grande sous-unité de l'ARN polymérase II a un domaine C-terminal ("carboxy-terminal domain": CTD) qui consiste en une répétition d'une séquence consensus de 7 acides aminés. Cette séquence répétitive est propre à l'ARN polymérase II. Chez les mammifères, on démombre environ 50 répétitions de cette séquence consensus. La portion CTD peut être hautement phosphorylée sur des résidus sérine ou thréonine. Dans ce chapitre nous nous intéresserons essentiellement à la synthèse des transcrits primaires des gènes des eucaryotes par l'ARN polymérase II.
Structure des gènes chez les eucaryotes: les exons et les introns.
Chez les eucaryotes, les gènes présentent une structure discontinue, comportant des exons et des introns. Les exons sont par définition des séquences d’ADN qui seront traduites en protéines (on dit aussi qu’elles seront exprimées). Les introns sont par définition des séquences d’ADN intercalées entre les exons.

Les promoteurs
- Tout d’abord, la boîte TATA.
Elle est située à environ -25 paires de bases de l’origine de la transcription. C’est une séquence de six nucléotides riche en A et T. La séquence dite consensus (statistiquement la plus rencontrée) est TATAAA. Une mutation dans cette boîte altère fortement la transcription. Cette boîte fixe un facteur général de transcription appelé TFIID (TF: facteur de transcription; II pour l’ARN polymérase II). Ce facteur est absolument nécessaire pour l’initiation de la transcription. Puis en remontant en amont du site d’initiation, on trouve successivement:
- La boîte GC (située le plus souvent dans la région entre -110 et -40). Elle peut se présenter sous forme d’hexanucléotides: 5’-GGGCGG-3’. Le motif riche en bases G et C peut être répété plusieurs fois.
- La boîte CCAAT (souvent située dans la région entre -120 et -80). Cette boîte peut être située avant ou après une boîte GC ou même entre deux boîtes GC.
L’unité de transcription comporte une origine le point de départ de la transcription (ou site +1) et un point de fin de la transcription (séquence de terminaison de la transcription). En d’autres termes, une unité de transcription va de la première base transcrite à la dernière base transcrite. L’ARN qui lui correspond s’appelle le transcrit primaire ou pré-mARN. Il comprend non seulement les régions codantes ou exons, mais aussi les introns et les portions 5’ et 3’ non traduites (régions 5’-UTR et 3’-UTR, « UTR= untranslated regions »).
Date de dernière mise à jour : 22/03/2018
Commentaires
-

1 BAZILE Le 02/04/2013

Ajouter un commentaire