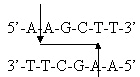- Accueil
- D.Sciences biologiques
- II.Biologie moléculaire COMPLET
- bio moléculaire 3 bis
bio moléculaire 3 bis
III.3.3 Structure tertiaire
Rappel : La chromatine est constituée d’ADN et de protéines.
Ces protéines vont imposer de nombreuse contraintes à la double hélice
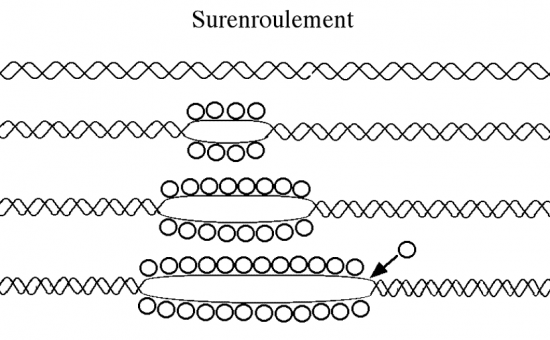
Conséquences : réactions de l’ADN qui se traduira par l’apparition de surenroulements .
Lors de la transcrition ou de la réplication, l’hélice du DNA est ouverte par une topoisomérase (hélicase) qui permet aux enzymes l’accès au brin modèle.
• Le fait de séparer les bases complémentaires et les deux brins de la double hélice sur plusieurs dizaines de nucléotides se traduit par un resserrement des tours d’hélice de part et d’autre de la boucle ainsi ouverte.
• Ce surenroulement nécessite l’intervention d’une topoisomérase qui va desserrer les tours d’hélice en coupant un brin ou les deux brins du DNA, puis en les faisant tourner l’un autour de l’autre jusqu’à revenir à 10 nucléotides par tour d’hélice.
• Des protéines de stabilisation du DNA simple brin viennent se fixer sur la partie déroulée pour protéger la molécule.
III.3.3.1 Cas des ADN circulaires
ADN viraux, mitochondriaux, des plasmides, des chloroplastes.
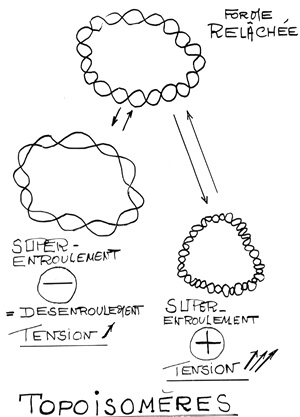
Deux molécules d’ADN présentant la même séquence de bases peuvent être différentes par le nombre d’enlacement, c’à d le nombre de tours que fait un brin autour de l’autre .
Les deux molécules porteront le nom de topoisomères.
Différents états des topoisomères
- Etat relaché : par conséquent contrainte minimum et stabilité de conformation
- Etats surenroulés : surenroulé + : plus de tours
- surenroulé - : moins de tours
Le surenroulement positif ou négatif exerce une contrainte sur la molécule qui donnera un vrillage.
L’obtention d’un ADN surenroulé nécessite un apport d’énergie, ce qui est normal puis que l’on passe d’un état sans tension à un état sous tension.
Les topoisomérases
Les topoisomérases sont des enzymes qui peuvent modifier le nombre d’enlacements
On en distingue les deux grands types :
Les topoisomérases I sont capables de couper transitoirement et de resouder un seul brin d’ADN double brin. Certaines topoisomérases de type I peuvent agir soit sur de l’ADN avec superenroulement positif ou négatif, d’autres agissent uniquement sur l’ADN avec superenroulement négatif.
Les topoisomérases II coupent de manière transitoire les deux brins de l’ADN, puis les ressoudent, c’est le cas de la gyrase bactérienne. Cette enzyme permet de désenrouler l’ADN.
Conséquences de l’action des topoisomérases
- L’ADN surenroulé est plus compact que son homologue relaché.
- Les topoisomérases favorisent la réplication, la transcription ou encore la recombinaison avec avec d’autres segments d’ADN. En effet, de nombreuses protéines intervenant dans ces mécanismes ne se lieront à l’ADN que s’il est surenroulé -, c’est à dire désenroulé .
Exemple : exemple d’un antibiotique sur colibacille : ce médicament bloque la girase, ce qui a pour conséquence d’empêcher les surenroulements et de bloquer la réplication de ces bactéries.
III.3.3.2 Cas des ADN linéaires ( cas des Eucaryotes)
Dans ce cas, il y a problème d’encombrement . En effet, le diamètre du noyau n’excède pas 1/100 ème de millimètre et la longueur de la molécule d’ADN est de l’ordre de grandeur du mètre, cet ADN étant ancré à la membrane nucléaire.
La solution à ce dilemme est la compaction de l’ADN .
Cette compaction donne naissance à la superhélice d’ADN
Il faut également assurer une certaine protextion aux brins d'ADN
Le DNA a besoin d’être protégé par des protéines lorsqu’il n’est pas utilisé comme modèle pour l’expression des gènes ou la réplication.
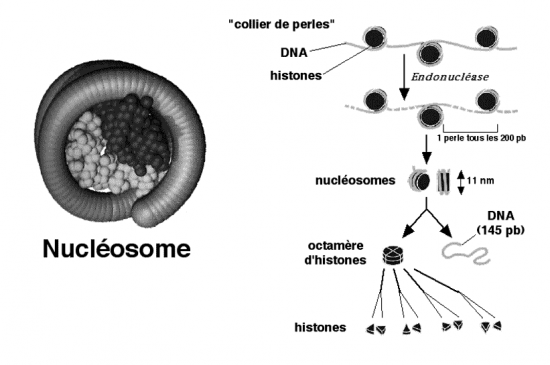
• Cette protection se fait par enroulement autour de protéines basiques (cationiques) capables de se lier avec le DNA qui est un polyanion. Des octamères d’histones sont au centre de particules qu’on trouve tous les 200 nucléotides et autour desquels le DNA s’enroule. La structure évoque un « collier de perles ».
• Le DNA ainsi lié aux histones est protégé contre l’action des enzymes. Une endonucléase peut digérer le DNA entre les « perles » et détacher des particules de 11 nm de diamètre appelées nucléosomes.
• Chaque nucléosome est constitué d’un fragment de DNA de 145 paires de nucléotides et de huit molécules d’histones.
Etude de la super hélice d’ADN
Les protéines accompagnant l’ADN dans la chromatine font partie de deux groupes
1. Les protéines histoniques
Ce sont des protéines basiques ou Histones (HP). Ces protéines sont stables et contiennent 20 % d’acides aminés basiques ( arginine, lysine).
Ces acides aminés sont regroupés du coté de la terminaison amine de la protéine , ce qui confère à cette partie un renforcement de charges +.
Il y a donc possibilité d’établissement de liaisons ioniques avec la molécule d’ADN.
Ces histones peuvent subir des modifications covalentes réversibles.
Résultat = changements de charges de ces protéines.
Exemple : la lysine peut subir une acétylation ( charge - )
La sérine peut subir une phosphorylation ( charge - )
Les rôles physiologiques de ces protéines peuvent ainsi être modifiés.
Hypothèse récente
Il a été découvert récemment que les histones oscillaient entre des états d'acétylation différents. Les histones acétylases dissocient la chromatine et permettent l'activation de la transcription. Les histones deacétylases referment la chromatine et bloquent toute activité.
Mode d'Action des Histones Acétylases
L'acétylation des histones se fait sur la partie NH2 terminale. Elle neutralise la charge positive de la partie N terminale des histones. Des modifications électrostatiques expliquent alors le changement de conformation du nucléosome, les charges négatives de l'ADN étant moins attirées par la partie Nter des histones maintenant neutre. Il est probable que chaque histone soit modifiée de manière différente et spécifique et que cela implique des réponses différentes, mais cet aspect reste pour l'instant mal connu.
Facteurs de Transcription et Histone Acétylases
Une des étapes essentielle de l'activation d'un facteur de transcription est donc son association avec une acétylase (activateur) ou déacetylase (répresseur). Ce co-facteur va moduler l'accessibilité des promoteurs et permettre ou non la régulation.
Importance des Histones Acetylases
La régulation par acétylation/déacetylation semble être commune a tous les facteurs de transcription. L'association avec une histone acétylase (ou déacetylase) constitue une étape initiale et obligatoire de l'activation transcriptionnelle.
Les différentes histones
H1 H2A H2B H3 H4
H2A + H2B se groupent pour former un dimère
2 H3 + 2 H4 se groupent pour former un tétramère.
Le tétramère entouré de deux dimères donne un octamère de 11nm de diamètre et de 5,5 nm de hauteur.
En présence de ce bloc d’histones, l’ADN va être contraint d’ adopter une conformation super hélicoïdale, le tout formant un nucléosome.
Le nucléosome est constitué :
- D’une bobine d’histone
- D’un tour ¾ d’une super hélice gauche soit 146 paires de nucléotides.
Deux nucléosomes sont séparés par un ADN bicaténaire d’environ 54 paires de nucléotides .
Rappels : voir biologie moléculaire 1 et 2 de cette catégorie
La fibre de chromatine peut être comparée à un collier de perles
Chaque perle étant un nucléosome avec son ADN nucléosomique , entre les nucléosomes, de l’ADN internucléosomique ( 20 paires de bases. ).
Rôle de H1
Cette histone se place à l’entrée et à la sortie de l’ADN du nucléosome, son rôle est de provoquer un reploiement de la fibre en solénoïde, une compaction de l’ADN
Le DNA qui entoure chaque nucléosome et le relie en « collier de perles » aux nucléosomes suivants, forme la trame d’une structure hélicoïdale qui enroule les colliers de perles sur euxmêmes.
On décrit aussi cette structure comme un solénoïde.• Le diamètre de cette hélice est de 30 nm et forme une grande partie de la chromatine dite
« compactée » où le DNA n’est pas accessible.
• Toutefois, des séquences reconnues spécifiquement par des protéines de liaison au DNA interrompentde place en place cette structure compacte.
2. Les protéines non histoniques (NHP)
Ce sont des protéines hétérogènes, métaboliquement instables , à renouvellement rapide. Elles sont présentes en petites quantités et mal connues.
Parmi elles, on trouve les GRP ( gene regulatory protein) . Ces protéines régulatrices de gènes sont essentiellement des activateurs chez les Eucaryotes.
Elles viennent se placer à proximité du site de l’ARN polymérase.Elles facilitent la liaison de cette enzyme avec l’ADN et activent de la sorte la transcription.
Voir la régulation génétique.
Conclusion : Ces protéines qui ont une affinité variable pour l’ADN permettent de comprendre l’expression sélective de l’information génétique.
Expression sélective de l’information :
- Variable d’un type de cellule à l’autre
- Variable en fonction des besoins physiologiques d’une même cellule.
Il convient de ne pas oublier que dans le noyau de chaque cellule d’un organisme existe un génome complet
III.4 Notions sur l’étude des séquences d’ADN
Quelles sont les différences entre deux molécules d’ADN ?
Les différences portent uniquement sur les séquences de bases.
Comment déterminer ces séquences de base ?
1. Découverte d’enzymes particulières en 1978, des endonucléases de restriction.
2. Ces enzymes sont capables de couper l’ADN en des endroits bien précis.
3. Possibilité d’analyser les fragments ainsi obtenus ( partie non développée dans ces notes).
Les enzymes de restriction
On connaît actuellement environ 200 enzymes de ce type , chacune reconnaît une séquence de nucléotides qui lui est spécifique.
Les enzymes ont été classées en 3 groupes. Les enzymes de type I et de type III ont à la fois l'activité de modification (méthylation) et l'activité de restriction (coupure) dépendante de l'ATP.
Les enzymes de type I et III se distinguent par la manière de couper l'ADN. Elles ne sont pas très utilisées en génie génétique.
Les enzymes de restriction de type II appartiennent à des systèmes binaires composés d'une endonucléase de restriction qui clive une séquence spécifique de nucléotides et d'une méthylase distincte qui modifie la même séquence de reconnaissance.
Les enzymes de restriction coupent les deux brins d'une molécule d'ADN au niveau de séquences de reconnaissance particulières, typiquement 4 à 8 nucléotides avec un axe de symétrie d'ordre 2 (palindrome).
Ceci génère, pour chaque brin, deux extrémités, l'une ayant un groupement 3'-OH, l'autre un groupement 5'-phosphate. Environ 150 enzymes de restriction ont été isolées à partir de plusieurs centaines de souches bactériennes.
Les noms des enzymes sont des abréviations des souches bactériennes à partir desquelles elles ont été isolées Un chiffre romain indique l'ordre de découverte des enzymes. Les enzymes les plus utilisées au laboratoire sont celles qui reconnaissent un site de quatre ou de six nucléotides.
Séquences reconnues
Comme spécifié plus haut, les séquences reconnues sont nommées palindromes
( ex radar).
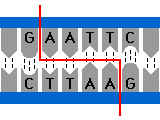
Exemple :
Brin 1 5’CCA GAATTC CAT 3’
Brin 2 3’GGT CTTAAG GTA 5’
Autre exemple avec endroit de coupure, l’enzyme de restriction Hind III qui reconnaît la séquence spécifique (et palindromique) suivante:
Le palindrome reconnu a de 4 à 8 paires de bases.
Origine de ces enzymes
C’est une origine bactérienne. Chez la bactérie, ces endonucléases auraient pour rôle de déparasiter, d’enlever les séquences de bases virales.
Les enzymes de restriction agiraient comme de véritables sécateurs.
Caractéristiques des coupures
Deux types de coupures possibles :
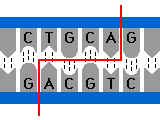
1. Coupures franches : c’a d coupure au milieu du palindrome.
Exemple :
2. Coupures en bouts collants :
Exemple
Remarques
1. L’enzyme de restriction coupera chaque fois qu’elle rencontrera son palindrome spécifique.
2. N’y a-t-il pas un danger pour la bactérie que ses enzymes ne coupent son propre ADN ? Car sur son ADN se trouvent aussi des palindromes.
Pour éviter ce désagrément , la bactérie va modifier ses propres palindromes : ce qui est réalisé par des enzymes appelées méthylases de modification ou enzymes de méthylation.
L’ADN bactérien présente des sites de restriction susceptibles d’être repérés par les enzymes de restriction que possèdent la bactérie.
Pour éviter une auto-destruction, les enzymes de modification de l’ADN bactérien interviennent.
La méthylation de la cytosine (sur le carbone 5) ou de l’adénine (sur l’azote 6) appartenant à des sites de restriction aboutit à une inactivation de l’enzyme de restriction correspondante.
Cette méthylation peut se réaliser sur une base ou sur plusieurs bases appartenant au site de restriction. Les méthylases bactériennes sont très spécifiques. Prenons l’exemple de l’enzyme de restriction Hind III qui reconnaît la séquence spécifique (et palindromique) suivante:
La méthylation de l’adénine (représentée sur le schéma associée avec un cercle) aboutit à une absence de reconnaissance de ce site spécifique par l’enzyme Hind III et donc à une absence de coupure enzymatique:
5’-Å-A-G-C-T-T-3’
PRECEDENT
SUIVANT
Date de dernière mise à jour : 17/06/2020
Ajouter un commentaire